Kolejna środa nadeszła, a to oznacza nową porcję przykładów. Dzisiaj poznamy różne sposoby walidacji krzyżowej. Nie przedłużając wstępu zapraszam do lektury.
Prosta walidacja
Dane
Z poprzednich przykładów na pewno już wiemy jak wygenerować lub wczytać dane. Dla szybkiego przypomnienia poniżej fragment kodu generowania danych. Wprowadzona jest drobna zmiana i dodajemy parametr weights, o którym dowiemy się więcej w dalszej części.
from sklearn.datasets import make_classification
X, y = datasets.make_classification(
weights=[0.8,0.2],
n_samples=100,
n_features=2,
n_informative=1,
n_repeated=0,
n_redundant=0,
flip_y=.05,
random_state=1234,
n_clusters_per_class=1
)
Podział danych
Wykonanie walidacji modelu wymaga od nas podziału danych na zbiór uczący i zbiór testowy. Trenowanie i testowanie modelu na takich samym danych jest bez sensu, a wyniki uzyskane w ten sposób nie pozwolą nam określić jakości naszego klasyfikatora. Najprostszym przykładem jest skorzystanie z funkcji train_test_split(), która pozwoli na wykonanie pojedynczego podziału naszego zbioru danych. Parametr test_size ustawiony na wartość .30 oznacza, że 30% losowo wybranych próbek ze zbioru zostanie przydzielona do zbioru testowego , a 70% do zbioru uczącego. Pamiętamy o random_state.
from sklearn.model_selection import train_test_split
X_train, X_test, y_train, y_test = train_test_split(
X, y,
test_size=.30,
random_state=1234
)
Budowa i testowanie modelu
Posiadając zbiór uczący możemy zbudować model. Klasyfikator wykorzystany do tego to znany z poprzedniego przykładu Naiwny klasyfikator bayesowski oparty na rozkładzie normalnym zwanym też rozkładem Gaussa. Zrobimy to za pomocą metody fit(), która jako argumenty przyjmuje atrybuty X_train i etykiety y_train zbioru uczącego. Kolejnym krokiem jest dokonanie predykcji na zbiorze testowym. Wykonuje się to za pomocą metody predict(), która jako argument przyjmuje atrybuty uczącego zbioru danych X_test. Zwracany jest zbiór przewidywanych etykiet predict , który jest odpowiedzią naszego wcześniej wyuczonego modelu. Ostatnim etapem jest wyliczenie jakości na podstawie wybranej metryki, w tym wypadku dokładność accuracy_score.
from sklearn.naive_bayes import GaussianNB
from sklearn.metrics import accuracy_score
clf = GaussianNB()
clf.fit(X_train, y_train)
predict = clf.predict(X_test)
score = accuracy_score(y_test, predict)
print("Accuracy score:\n%.2f" % score)
Jakość jaką otrzymaliśmy jest dość wysoka, ale nie oznacza to, że jest to wynik nas satysfakcjonujący.
Accuracy score: 0.900
Walidacja krzyżowa
K-krotna walidacja krzyżowa
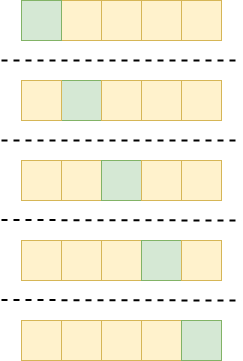
Przechodzimy teraz do dużo lepszej metody walidacji - k-krotnej walidacji krzyżowej. Pozwoli to nam na wyznaczenie jakości modelu w najczęściej stosowany sposób. Główną ideą jest podział naszego zbioru na K równych części. Następnie kolejno każda z tych części używana jest jako zbiór testowy, a pozostałe jako zbiór uczący. Proces ten wykonywany jest K razy i za każdym razem budujemy nowy model od nowa. Następnie uzyskane rezultaty (np. dokładność) są uśrednianie lub w jakiś inny sposób agregowane w jeden wynik. Poniżej wizualizacja podziału dla 5-krotnej walidacji krzyżowej. Całość to w pewnym uproszczeniu nasz zbiór danych podzielony na 5 losowych części. Każdy zielony kwadracik oznacza część wybraną jako podzbiór testowy , a żółte oznaczają podzbiór uczący. W sumie mamy 5 iteracji.
W praktyce wygląda to w następujący sposób. Najpierw po zaimportowaniu odpowiednich składników tworzymy obiekt kf klasy KFold z ustalonym parametrem liczby części n_splits. W tym wypadku ustalamy wartość 5. Pamiętajmy jeszcze o parametrze shuffle ustawionym na wartość True co oznacza, że dane zostaną przetasowane przed podziałem oraz o ustawieniu parametru random_state. Następnie tworzymy sobie pustą listę scores, na której będziemy później umieszczać uzyskaną dokładność na poszczególnych podzbiorach testowych.
from sklearn.model_selection import KFold
kf = KFold(n_splits=5, shuffle=True, random_state=1234)
scores = []
W kolejnym kroku wykorzystujemy metodę split z argumentem X, która zwróci nam numery indeksów, próbek wybranych i podzielonych na podzbiory uczące oraz podzbiory testowe. Odpowiednio train_index i test_index. Mechanizm pętli pozwala nam na iteracyjne wybieranie poszczególnych podziałów. Wewnątrz pętli dokonujemy już faktycznego podziału na atrybuty uczące X_train = X[train_index] i testowe X-test = X[test_index]. To samo dotyczy etykiet y_train, y_test = y[train_index], y[test_index]. Mając już tak podzielony zbiór danych możemy zbudować model, dokonać predykcji i obliczyć dokładność klasyfikacji, a uzyskany wynik zamieścić na wcześniej przygotowanej liście scores.
for train_index, test_index in kf.split(X):
X_train, X_test = X[train_index], X[test_index]
y_train, y_test = y[train_index], y[test_index]
clf.fit(X_train, y_train)
predict = clf.predict(X_test)
scores.append(accuracy_score(y_test, predict))
W ostatnim kroku uśredniamy np.mean() uzyskane wyniki oraz liczymy standardowe odchylenie np.std(). Dokonujemy tego za pomocą funkcji zawartych w bibliotece numpy.
import numpy as np
mean_score = np.mean(scores)
std_score = np.std(scores)
print("Accuracy score: %.3f (%.3f)" % (mean_score, std_score))
Uzyskany przez nas wynik to:
Accuracy score: 0.880 (0.051)

-
Czy to oznacza, że pogorszyliśmy jakość naszego modelu?
-
Stanowcze nie!
Na pewno poprawiliśmy jakość naszego badania. Może się też zdarzyć, że zmierzona dokładność ulegnie zmianie, ale nie zawsze będzie się pogarszać.
Stratyfikowana walidacja krzyżowa
Niejednokrotnie możemy się spotkać z danymi gdzie liczba obiektów danej klasy jest liczniejsza od pozostałych klas. W przypadku niewielkich różnic nie ma to dużego wpływu na wynik i przeprowadzanie badania. Jednak duże różnice stają się pewnym utrudnieniem. Nazywane jest to niezbalansowaniem danych. Wiąże się to z zastosowaniem odpowiedniego podejścia podczas klasyfikacji i przeprowadzania eksperymentów. Za pomocą prostej komendy możemy wypisać sobie wszystkie etykiety:
print(y)
Co powoduje wypisanie:
[0 0 0 0 0 0 0 0 0 0 1 0 0 0 0 0 1 1 0 0 0 0 1 1 0 0 1 0 0 0 0 0 0 0 0 0 0
0 0 0 0 1 0 0 0 1 0 0 0 1 1 0 0 0 1 0 0 0 0 0 1 0 1 0 0 0 1 0 0 0 0 1 0 0
0 1 0 1 1 0 1 0 1 0 1 0 1 0 0 0 0 0 0 0 1 0 1 0 0 0]
Na pierwszy rzut oka wygląda, że jest więcej etykiet klasy “0” niż klasy “1”. Możemy sobie oszczędzić ręcznego liczenia i wykorzystać jedną z funkcji biblioteki numpy do sprawdzenia ile dokładnie jest poszczególnych wartości.
print(np.unique(y, return_counts=True))
Wynikiem czego jest:
(array([0, 1]), array([76, 24]))
Oznacza to, że w naszym zbiorze danych klas oznaczonych etykietą “0” jest dokładnie 76, a oznaczonych etykietą “1” jest 24. Nasze dane również są niezbalansowane. Ten współczynnik pomiędzy liczbą etykiet klas nazywamy poziomem niezbalansowania. Odpowiedzialny za to jest parametr generatora weights=[0.8,0.2] , za pomocą którego ustalamy jaki będzie udział poszczególnych klas w generowanych danych. Walidacja krzyżowa dokonuje losowego podziału na pozdbiory, co może zaburzać ten rozkład. W tym momencie bardzo przydatna okazuje stratyfikowana walidacja krzyżowa, która podczas podziału na podzbiory zachowuje oryginalny lub zbliżony do oryginalnego poziom niezbalansowania.
from sklearn.model_selection import StratifiedKFold
skf = StratifiedKFold(n_splits=5, shuffle=True, random_state=1234)
scores = []
for train_index, test_index in skf.split(X,y):
X_train, X_test = X[train_index], X[test_index]
y_train, y_test = y[train_index], y[test_index]
clf.fit(X_train, y_train)
predict = clf.predict(X_test)
scores.append(accuracy_score(y_test, predict))
mean_score = np.mean(scores)
std_score = np.std(scores)
print("Accuracy score: %.3f (%.3f)" % (mean_score, std_score))
Cała procedura odbywa się w podobny sposób jak przy zwykłej k-krotnej walidacji krzyżowej. Wykorzystujemy tylko inną klasę w tym wypadku StratifiedKFold oraz w metodzie split podajemy etykiety danych y, tak żeby walidator wyciągnął informacje o poziomie niezbalansowania klas. Na koniec sprawdzamy dokładność.
Accuracy score: 0.860 (0.066)
Wynik niższy, ale badania jeszcze bardziej zyskały na jakości, a my jesteśmy bogatsi o nową wiedzę.
Wielokrotna walidacja krzyżowa
Jeszcze innym podejściem jest wielokrotna walidacja krzyżowa. Głównie założenie tego sposobu jest takie, że dokonujemy kilku powtórzeń podziału na podzbiory. Innymi słowy jest to pewne zautomatyzowanie kilku krotnego procesu walidacji krzyżowej i obliczanie uśrednionej wartości z tych wszystkich podzbiorów. Praktycznie kod programu wygląda prawie identycznie jak w przypadku normalnej walidacji, różnica jest tylko w użyciu innej klasy RepeatedKFold oraz ustaleniu dodatkowego parametru n_repeats. W poniższym przykładzie wykonamy 5-krotną walidację krzyżową z 10-cioma powtórzeniami. W pewnym uproszeniu można powiedzieć, że robimy walidację krzyżową 10x5 (dziesięć na pięć).
from sklearn.model_selection import RepeatedKFold
rkf = RepeatedKFold(n_splits=5, n_repeats=10, random_state=1234)
scores = []
for train_index, test_index in rkf.split(X):
X_train, X_test = X[train_index], X[test_index]
y_train, y_test = y[train_index], y[test_index]
clf.fit(X_train, y_train)
predict = clf.predict(X_test)
scores.append(accuracy_score(y_test, predict))
mean_score = np.mean(scores)
std_score = np.std(scores)
print("Accuracy score: %.3f (%.3f)" % (mean_score, std_score))
Istnieje również stratyfikowana wielokrotna walidacja krzyżowa. Poniżej przykładowy kod programu.
from sklearn.model_selection import RepeatedStratifiedKFold
rskf = RepeatedStratifiedKFold(n_splits=5, n_repeats=10, random_state=1234)
scores = []
for train_index, test_index in rskf.split(X,y):
X_train, X_test = X[train_index], X[test_index]
y_train, y_test = y[train_index], y[test_index]
clf.fit(X_train, y_train)
predict = clf.predict(X_test)
scores.append(accuracy_score(y_test, predict))
mean_score = np.mean(scores)
std_score = np.std(scores)
print("Accuracy score: %.3f (%.3f)" % (mean_score, std_score))
Leave-one-out
Ostatnim już dzisiaj przykładem walidacji krzyżowej jest tak leave-one-out. Jest to bardzo szczególna odmiana k-krotnej walidacji krzyżowej, gdzie zbiór danych jest dzielona na podzbiory, zawierające tylko po jednym obiekcie danych. Podobnie jak w poprzednich walidacjach krzyżowych za każdym razem jeden podzbiór (tutaj jeden obiekt) używany jest do testowania, a pozostałe do uczenia. Tego typu metoda stosowana jest najczęściej dla małych zbiorów danych. Od praktycznej strony zastosowanie odbywa się w dość analogiczny sposób jak pozostałe dzisiejsze przykłady.
from sklearn.model_selection import LeaveOneOut
loo = LeaveOneOut()
for train_index, test_index in loo.split(X):
X_train, X_test = X[train_index], X[test_index]
y_train, y_test = y[train_index], y[test_index]
clf.fit(X_train, y_train)
predict = clf.predict(X_test)
scores.append(accuracy_score(y_test, predict))
mean_score = np.mean(scores)
std_score = np.std(scores)
print("Accuracy score: %.3f (%.3f)" % (mean_score, std_score))
To by było dzisiaj na tyle. Pełni nowej wiedzy możemy teraz z czystym sumieniem odejść od komputera i w tak piękną pogodę z pełną odpowiedzialnością za los naszego świata posiedzieć sobie w domu.